Design Racional de Fármacos Assistido por Computador - CADD
Desenvolvimento de agonista do receptor tireoidiano alfa para estimular regeneração cardíaca utilizando técnicas de biologia computacional e ensaios in vitro
As doenças cardiovasculares são as que mais matam no mundo, sendo as doenças isquêmicas as mais letais. Considerando o potencial regenerativo estimulado pelo Hormônio Tireoidiano no coração, utilizamos técnicas de biologia computacional para selecionar e otimizar moléculas agonistas do receptor tireoidiano alfa, por análises de Dinâmica Molecular, visando o desenvolvimento de potenciais fármacos para o tratamento de lesões cardíacas.
Membros deste projeto
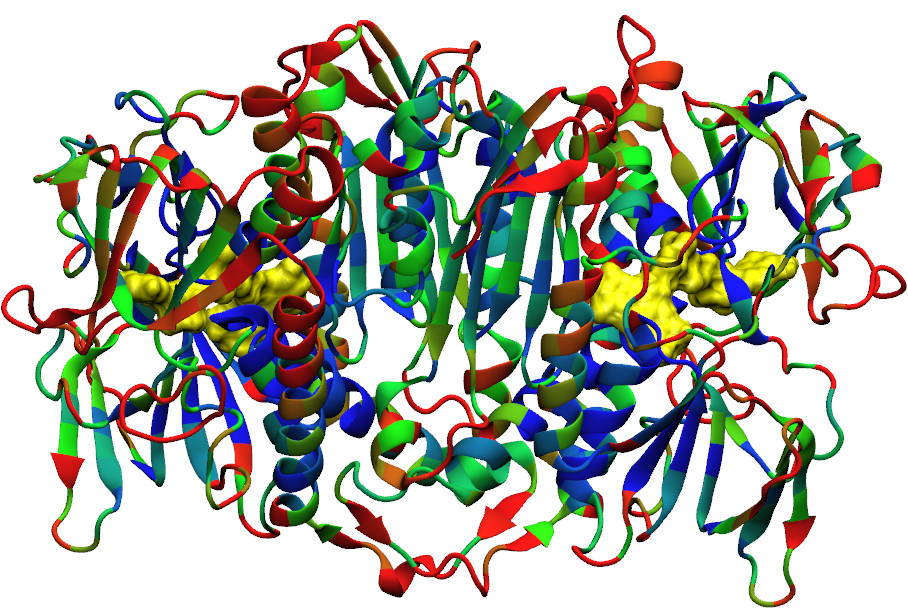
Desenho racional de inibidores ortostéricos e alostéricos de tripanotiona redutase assistido por técnicas computacionais.
O projeto visa a caracterização dos sítios ortostérico e alostéricos da enzima tripanotiona redutase por meio de emprego de técnica de dinâmica molecular em acordo com estudo de docagem e otimização de compostos líderes. Esse homodímero participa do controle de dano oxidativo exclusivamente em parasitos Tripanossomatídeos, configurando alvo de interesse no combate a protozoonoses tropicais endêmicas.
Membros deste projeto
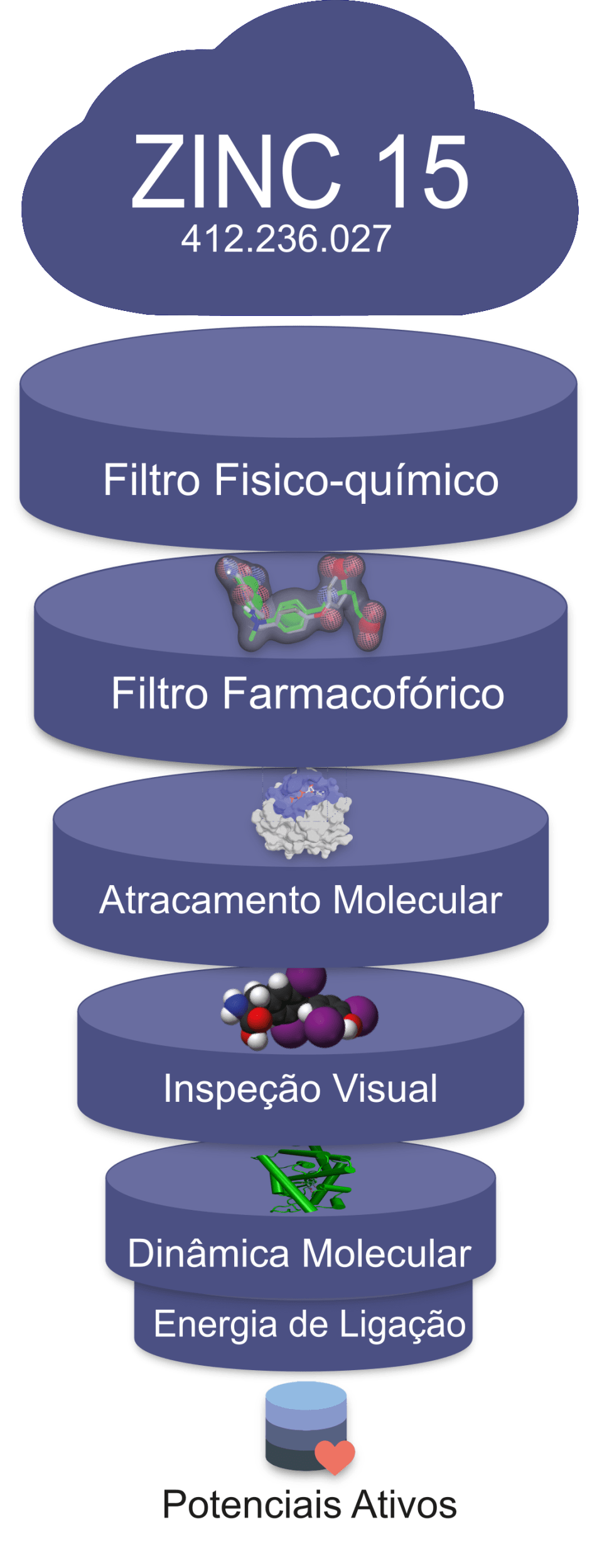
Inteligência Artificial em Triagem Virtual para criação de consenso
Quando a docagem molecular é realizada, existe a necessidade de se avaliar os resultados. Isso é feito através do uso de funções de pontuação, que são baseadas em diferentes abordagens. Entretanto, as vezes uma função de pontuação não retorna bons resultados em alguns casos, enquanto outras sim. Então, o uso da abordagem de consenso tem sido bem estudada visando resolver este problema. Uma vez que o comportamento das funções de pontuação não é linear entre cada complexos proteína-ligante, é complexo estabelecer uma relação simples entre cada função de pontuação, portanto, o uso de inteligência artificial (IA) foi escolhido, uma vez que a IA tem a capacidade de estimar implicitamente a relação entre dois ou mais elementos, mesmo essa relação sendo complexa. Baseado nesta ideia, este projeto tem como meta o desenvolvimento, implementação e validação de um sistema de docagem molecular que faça uso da IA a fim de se auxiliar o processo de descobrimento de fármacos. O pacote desenvolvido neste projeto também tem como meta, permitir a sua importação separadamente dentro de um script Python3, permitindo ao usuário executar a automação do processo de docagem molecular.
Membros deste projeto
Computational Modeling of ZIKV MTase NS5-SAH Complex
A infecção causada pelo Vírus Zika (ZIKV) tornou-se uma emergência de saúde pública de preocupação internacional, principalmente devido às desordens neurológicas associadas à infecção viral, como por exemplo a Síndrome de Guillain-Barré e microcefalia em fetos e recém-nascidos. Embora não haja vacina disponível, tratamentos alternativos têm sido investigados, buscando como alvo proteínas essenciais para a replicação viral. Dentre estas proteínas, a proteína não-estrutural 5 (NS5) constitui-se como um importante alvo farmacológico sendo a mais conservada entre os flavivírus, contendo um domínio metil-transferase (MTase) na região N-Terminal e um domínio RNA polimerase dependente RNA (RdRp) na porção C-Terminal. A porção MTase contém o cofator S-adenosil-metionina (SAM), responsável por transferir um grupamento metil, formando o subproduto S-adenosil-homocisteína (SAH). A MTase é responsável por modificar o RNA viral e impedir que o mesmo seja reconhecido pelo sistema imune do hospedeiro. Sendo assim, com a inibição da função da MTase, o vírus torna-se mais vulnerável, o que torna a NS5 MTase um alvo importante para o desenvolvimento de terapias antivirais. O objetivo do presente trabalho é mapear as interações da NS5 MTase complexada ao subproduto SAH, sugerindo um inibidor a partir de simulações usando ferramentas de biologia computacional.
Membros deste projeto
Otimização de inibidores alostéricos contra a protease principal da SARS-CoV-2
…







